NUCLEO CELULAR
Característico de las células eucariontes, excepto en glóbulos rojos (viven 120 días y son reciclados).
Su tamaño y ubicación son variables. En la mayoría de los tipos celulares está en el centro.
Almacena la información genética en el ADN.
Regulación de la expresión genética, convierte la información genética en proteí
Ejecutar, dirigir y regular las actividades citoplasmáticas, a través de las proteí
División celular, cuando una célula se divide tiene como objetivo compartir su ADN, por esto se duplica y luego se divide.
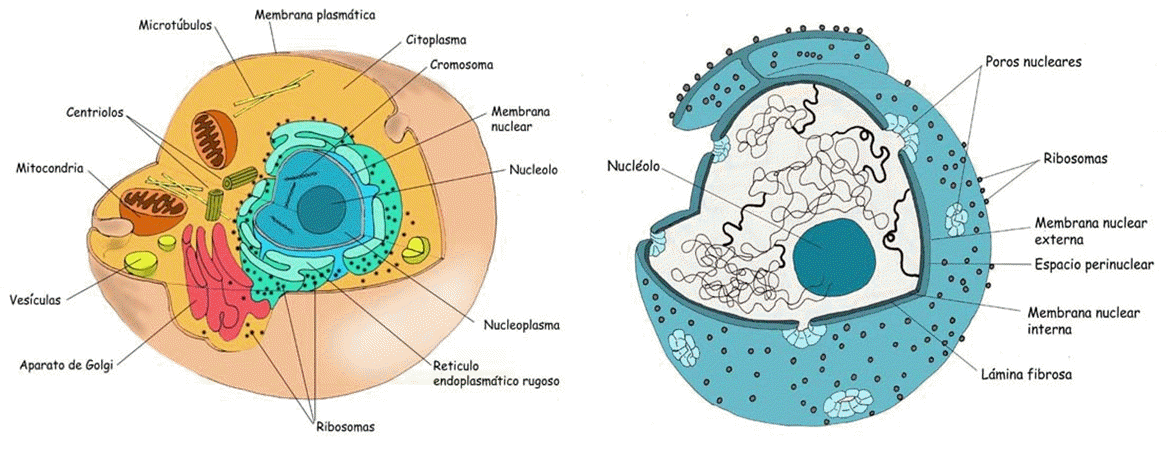
ESTRUCTURA: rodeado por envoltura nuclear, doble membrana separada por numerosos poros nucleares que actúan como una compuerta selectiva que permite el ingreso de ciertas proteínas desde el citoplasma, y también la salida de los distintos ARN y sus proteínas asociadas.
La envoltura nuclear esta sostenida desde el exterior por una red de filamentos intermedios dependientes del citoesqueleto. Y la lámina nuclear provee soporte interno.
En el nucleoplasma están disueltos los solutos del núcleo y la matriz nuclear que les da soporte a los cromosomas y a los grandes complejos proteicos que intervienen en la replicación y transcripción del ADN.
Los cromosomas ocupan lugares específicos. Los genes que codifican productos relacionados, aunque estén en distintos cromosomas, pueden estar cerca en el núcleo interfásico.
Los genes del ARNr están todos juntos y encerrados en el nucléolo, donde se sintetizan, procesan y ensamblan los ARNr. Y esa división física asegura que los ARNr sean correctamente ensamblados dentro de las subunidades ribosomales.
En el núcleo, los genes transcripcionalmente activos están ubicados centralmente, separados de los inactivos que se encuentran cerca de la envoltura nuclear.
LA ENVOLTURA NUCLEAR
Formada por dos membranas interrumpidas por poros nucleares y por la lámina nuclear. Las membranas están separadas por el espacio perinuclear (10 a 50 nm). La membrana externa está en contacto con el citoplasma y tiene ribosomas adheridos, que sintetizan las proteínas que se vuelcan al espacio perinuclear, el cual se continua con el REG. La membrana interna posee proteínas integrales propias que se unen a la lámina nuclear y a los cromosomas.
La envoltura nuclear deriva del REG y su aparición permitió que los eucariontes aíslen los procesos genéticos principales, como la autoduplicación de ADN o síntesis de ARN.
La lamina nuclear es una capa fibrosa (10 a 15nm) en la que apoya la membrana interna. Está formada por laminofilamentos (proteínas) que se unen a las proteínas integrales de la membrana. Le da sostén a la envoltura nuclear, y si se desensambla, la envoltura nuclear se desarma. Por ende, le da sostén al núcleo.
COMPLEJOS DE PORO NUCLEAR
Son estructuras proteicas que forman canales que atraviesan la envoltura nuclear. Su número es variable, incrementándose a medida que aumenta la actividad celular. Estos poros están porque regulan el tránsito de grandes sustancias (proteínas, ARN, ribosomas) entre en núcleo y el citosol
Formadopor:
8 columnas proteicasà forman las paredes laterales del poro
Un anillo externo.
Un anillo interno.
Proteínas de anclajeà fijan cada columna al espacio perinuclear.
Proteínas radiales.
Proteínas fibrilaresà en la cara nuclear convergen para formar una canasta.
Nucleoporinasà intervienen en el transporte de sustancias a través del poro.
Un poro central o abertura.
TRANSPORTE A TRAVES DEL CPN
Los CPN tienen uno o varios canales por los cuales difunden las pequeñas moléculas solubles en agua (transporte no regulado). Las moléculas más grandes son transportadas de forma activa, por lo que requieren energía y moléculas transportadorasàproteína “transbordadora”o cariotransportinaàimportinas y exportinas.
Se importan dentro del núcleo:
Las proteínas sintetizadas en el citoplasmaànecesarias para ensamblar los ribosomas.
Los factores de transcripciónà para la activación o inactivación de los genes.
Los factores de empalmeà necesarios en la maduración de los ribosomas.
Las moléculas y macromoléculas ensambladas y exportadas desde el núcleo al citoplasma incluyen:
Las subunidades ribosomales.
ARN de transferencia.
Factores de transcripciónà se devuelven al citoplasma para ser reutilizados.
Los CPN hacen de la envoltura nuclear una barrera selectiva entre el núcleo y el citoplasma. Las proteínas grandes para entrar al núcleo por el canal central tienen una señal de localización nuclear. Y los ARN salen del núcleo a través del poro con una proteína que tiene una señal nuclear de exportación.
CROMOSOMAS Y CROMATINA
El núcleo contiene los cromosomas de la célula, cada uno de ellos consiste en una molécula única de ADN con una cantidad equivalente de proteínas. El ADN con sus proteínas asociadas se denomina cromatina.
La mayoría de las proteínas de la cromatina consisten en muchas copias de 5 clases de histonas. Estas proteínas básicas cargadas positivamente se unen con los grupos fosfatos (cargados negativamente) del ADN. También hay gran variedad de proteínas no histónicas y RNP en la cromatina, la mayoría son factores de transcripciónà regulan que parte del ADN será transcripta en ARN.
NIVELES DE ORGANIZACIÓN DE LA CROMARINA
Heterocromatina o cromatina densaàen la periferia del núcleo, representa aprox. el 10% del total de la cromatina y es considerada transcripcionalmente inactiva. Presenta ADN que o bien carece de información genética, o bien tiene información genética pero no está disponible para la cé Zona más compactada.Heterocromatina constitutivaà mismo ADN para todas las células de un mismo individuo; tejido dependiente.Heterocromatina variableà varia de un tejido a otro.Eucromatina o cromatina laxaàmenos compactada, de localización central, su información está siempre disponible para la cé Se tiñe débilmente. Se presenta en dos estados:Eucromatina accesibleàdonde se encuentran los genes que se están transcribiendo.
El andamiaje o matriz nuclear se asocia a la cromatina a nivel de zonas altamente conservadasà SAR o MARà regiones de cientos de pares de bases ricos en residuos de adenina y timina, abundantes en la heterocromatina.
EL NUCLEOSOMA
Los nucleosomas son las unidades de enrollamiento de la cromatina. Están formados por un centro de histonas y a su alrededor 146 pares de bases del ADN se enrollan en dos vueltas. El empaquetamiento de los nucleosomas esta mediado por la quinta histona, la H1, que los conecta y actúa como banda de goma manteniéndolos juntos dentro de una misma cuerda.
El empaquetamiento de la cromatina permite contener 2 metros de ADN dentro del núcleo y también lo protege del ataque de nucleasas.
EL CROMOSOMA EUCARIOTA
Cada uno consiste en una molécula simple de ADN lineal, es decir, que tiene dos extremos.
La molécula de ADN de un cromosoma eucariota contiene:
Un conjunto lineal de genes que codifican para ARN y proteí
Muchas secuencias de ADN no codificante, el cual incluye:
Origen de replicación (ORI)àmúltiples secuencias señalizadorasàpara que se realice la duplicación del ADN en poco tiempo.
Centrómeroàsecuencias de ADN satélite repetidos miles de veces y condensados siendo parte de la heterocromatina. Es una limitación primaria localizada centralmente hacia los extremos de cada cromosoma
Telómerosà secuencias repetitivas en los extremos del cromosoma. Necesarios para la duplicación completa del cromosoma, los protegen de las nucleasas, evitan que los extremos del cromosoma se fusionen entre sí y facilitan su interacción con la envoltura nuclear.
La telomerasa es una ribonucleoproteínaretrotranscriptasa que sintetiza ADN a partir de un molde de ARN. Las células con telomerasa activa (células germinales, eucariotas unicelulares y células cancerosas) pueden compensar el acortamiento de los telómeros durante la duplicación de ADN.
La posición del centrómero permite diferenciar a los cromosomas. Al inicio de la división celular, los cromosomas duplicados se mantienen juntos por el centrómero y es común llamar cada parte del cromosoma duplicado cromátida hermana. Cada una de ellas es un cromosoma completo.
El cinetocoro es una estructura proteica en forma de disco que forma parte del centrómero y ayuda a separar las cromátidas hermanas.
La posición del centrómero determina el largo de los brazos del cromosoma; en base a esto se puede clasificar a los cromosomas:
Metacéntricos: el centrómero en el centroà brazos de igual longitud.
Submetacéntricos: centrómero alejado del centroà un par de brazos más corto que el otro.
Acrocéntricos: centrómero próximo a uno de los extremosà un par de brazos es casi inexistente.
Telecéntricos:centrómero en uno de los extremos.
Numero diploide (2n): número característico de pares de cromosomas homólogos que tienen todas las especies.
CARIOTIPO
Es una representación gráfica de los cromosomas presentes en el núcleo de una sola célula somática de un individuo. Cada uno de los cromosomas homólogos proviene de cada uno de los padres del individuo.
El cariotipo de la mujer tiene 23 pares de cromosomas homólogos: 22 pares de autosomas y 1 par de cromosomas sexuales, ambos “X”.
El cariotipo del hombre tiene los mismo 22 pares de autosomas y 1 par de cromosomas sexuales, pero un cromosoma de ese par es “X” y el otro “Y” (posee un gen que determina el sexo).
EL NUCLEOLO
Participa en la formación de subunidades ribosómicas, la síntesis y procesamiento de ARNr, y en la regulación del ciclo celular.
Es un conjunto de fibras de cromatina de distintos cromosomas.
Sus cromosomas son todos acrocéntricos y presentan organizadores nucleolares (NOR), donde están los genes que codifican ARNr.
Es una estructura simple carente de membrana.
Se diferencia en dos regiones:
Zona fibrilar central, formada por ADN ribosómico y ARNr naciente.
Zona granular perifé
Desaparecen en la mitosis y se reorganizan alrededor de los segmentos de ADNr.
NATURALEZA MOLECULAR DEL GEN Y DEL GENOMA
GEN
Concepto mendelianoàes una unidad informativa discreta, responsable de una característica transmisible. Dicha unidad informativa es un fragmento de ADN que se encuentra en un lugar determinado del cromosoma.
Los genes especifican la estructura de las proteínas individuales. Cada proteína consiste en una secuencia de aminoácidos típica, de la cual dependen sus propiedades. Al haber mutaciones (alteraciones en la secuencia de nucleótidos del ADN) surgen alteraciones en la secuencia de aminoácidos en las proteínasàla secuencia de ADN especifica, mediante algún código, la secuencia proteicaàun gen es una secuencia de ADN con la información necesaria para la síntesis de una proteína particular.
La información del ADN se expresa indirectamente a través de otras moléculas. El ADN dirige la síntesis de proteínas t éstas determinan las características físicas y químicas de las células.
Solo el ARNm es portador de información acerca de la secuencia de aminoácidos de una proteína; aunque todos los ARN son transcriptos del ADNà segunda definición del genàun gen es una secuencia de ADN transcripta que genera un producto con función celular especifica. Pero también existen regiones reguladoras de los genes que no se transcriben, y otras (intrones) que se transcriben, pero se eliminan sin cumplir ninguna función.
Funcionan como unidades de herencia.
GENOMA
Es el conjunto de genes de una especie.
ORGANIZACIÓN DEL GENOMA EN PROCARIONTES Y EUCARIONTES
|
Procarionte |
Eucarionte |
|
ADNà una única molécula circular. |
ADNà estructura lineal. Usualmente más de uno en su núcleo. |
|
"ADN desnudo"àno se asocia a histonas. |
ADN asociado a histonas (empaquetamiento de ADN). |
|
No tiene envoltura nuclear. Traducción y trascripción no están separados en espacio y tiempo. |
Transcripciónà en cromosomas en el compartimiento nuclear. Traducciónà citoplasma. Ambos separados espacial y temporalmente. |
|
Los ARNs no son sometidos a modificaciones postranscripcionales. |
Los ARNs experimentan en el núcleo un proceso de maduración previo a la traducción. |
|
Genomas mas pequeños. |
Genomasmásgrandes. |
|
Cada cromosoma tiene una sola copia de cualquier gen particular y prácticamente se expresa todo el ADN. |
Gran exceso de ADN "innecesario". |
COMPLEJIDAD DEL GENOMA EUCARIOTA
Tres tipos de secuencias en el ADN eucariota:
Altamente repetidas.
Medianamente repetidas.
No repetidas o de copia ú
Secuencias altamente repetidas. Orden de copias por genoma y constituyen casi el 10% del ADN de los vertebrados. Son secuencias cortas y se repiten en forma consecutiva y sin interrupció Se encuentran en la heterocromatina y no hay evidencia de que se expresen. Se divide en diferentes categorías:
ADN satélite. Secuencias cortas (de 5 a 100 pares de bases). Se repiten gran cantidad de veces formando grupos muy amplios. Ej: ADN de los centró
ADN minisatelite. Secuencias de 15 pares de bases, entre 1000 y 3000 repeticiones.
ADN microsatélite. De 2 a 5 pares de bases, en grupos de 100 copias.
Secuencias medianamente repetidas. Entre el 20 y 80% del ADN total. Secuencias de cientos de bases de longitud, y entre 50 y 100000 copias de cada secuencia. Se ubican dispersas a lo largo del genoma.
Secuencias con función codificadora. Las secuencias que codifican ARNt, ARNr y los genes de las histonas. Se ubican en serie.
Secuencias sin función codificadora. Corresponde a la mayor parte del ADN medianamente repetido. Son secuencias dispersas de dos tipos: ECIN (elementos cortos interpuestos) y ELIN (elementos largos interpuestos). De 500 a 1000 pares de bases.
Secuencias de copia ú Comprenden a las secuencias de nucleótidos que codifican proteínas.
PROCESO DE TRANSCRIPCION
La transcripción consiste en la síntesis de ARN a partir de un molde de ADN.
Presenta dos características principales:
Es selectivaà porque cuando se transcribe un gen no se transcribe la molécula de ADN entera, solo el pedacito que ocupa el gen.
Es asimétricaàsolo 1 de las 2 cadenas es la que funciona como molde (negativa o no codificante) para la transcripcióLa cadena no transcripta se denomina antimolde (positiva o codificante).
La transcripción involucra la participación de una enzima ARN polimerasa dependiente. Esta sintetiza una cadena de ARN cuya secuencia de bases viene determinada por el propio gen.
Primer paso de la transcripciónà unión de la enzima ARN polimerasa a una región del gen llamada promotor. Este es una secuencia de bases con alta afinidad por la enzimaàle proporciona su sitio de unión al ADN.También es una señal que indica cuando una cadena se transcribe. El promotor reconoce la ARN polimerasa y le indica donde comienza la región codificante y cuál es la cadena moldeà la ARN polimerasa lo primero que hace cuando entra al ADNes separar las cadenas para tener acceso a la cadena moldeàla doble hélice sufre un desenrollamiento y fusiónà la ARN polimerasa corta las uniones puente de hidrogeno entre las cadenas complementarias y va formando el ARN a medida que avanza, generando una burbuja de transcripción (tramo de aprox. 12 nucleótidos de longitud en el que las cadenas permanecen separadas)à la formación de la burbuja causa superenrollamiento de la doble hélice hacia el extremo 5’ del moldeà para evitar que el ADN se rompa por superenrollamiento, la topoisomerasacorta la doble hélice, la desenrolla y la vuelve a juntar.
La ARN polimerasa se desplaza sobre la cadena molde en dirección 3’=> 5’ (rio abajo) y la transcribe a partir del nucleótido que el promotor señala como punto de inicio de la transcripciónà este nucleótido se nombra +1 y los siguientes rio abajo sigue la numeración (+2,+3); partiendo en la dirección contraria, “rio arriba”, los nucleótidos se numeran -1,-2,-3, etc.
|
A |
T |
C |
G |
Desoxi. |
|
U |
A |
G |
C |
Ribo |
Cuando la cadena molde es separada en la burbuja de transcripción y expone sus bases, estas son reconocidas por la ARN polimerasa. A medida que la enzima lee la cadena, coloca junto a cada base de la misma el ribonucleótido trifosfato portador de la base complementaria. A los desoxirribonucleótidos de A, T, C y G se le aparean, respectivamente, los ribonucleótidos de U, A, G y C mediante puentes de hidrogeno.
Una vez ubicados los dos primeros ribonucleótidos, la enzima cataliza la formación del puente fosfodiéster entre ambos (grupo hidroxilo 3’ del primero y grupo fosfato 5’ del segundo), iniciándose la cadena de ARN. Un grupo pirofosfato del segundo es liberado como producto y separado rápidamente en dos fosfatos por la acción de una pirofosfatasa.Dicha ruptura es tan exergónica que son los mismos sustratos los que, por estar trifosfatos, aportan la energía necesaria para su polimerización.
Este procedimiento se repite tantas veces como nucleótidos contenga el molde, de tal manera que el ARN va creciendo antiparalelo a aquelà la dirección de la síntesis de ARN es 5’=> 3’.
La transcripción concluye cuando la ARN polimerasa alcanza una señal que actúa como señal de terminación.
El producto obtenido, un ARN transcripto primario, resulta una copia complementaria y antiparalela de una región del gen comprendida entre el punto de inicio y la señal de terminación. El transcripto primario repite la dirección y la secuencia que la cadena anti molde (excepto que lleva U en vez de T).
|
Requisitos de la transcripción: - Una molécula de ADN molde, con región promotora, que indica el inicio de la transcripcion, con secuencia de terminación. - Una enzima ARN polimerasa que: · Reconoce las secuencias señalizadoras, Abre la hélice, Lee el molde (3’=>5’), Reconoce y ubica los sustratos complementarios, Polimeriza los sustratos (5’=>3’). - Cofactores enzimáticos de la ARN polimerasa: Mg++ o Mn++. - Sustratos: UTP, ATP, GTP y CTP. - Una fuente de energía: los mismos sustratos. - Una pirofosfatasa. - Una topoisomerasa. |
TRANSCRIPCION EN PROCARIONTES
Las procariontes presentan un solo tipo de ARN polimerasa que sintetiza todos los ARNs de la célula. Está constituida por 5 tipos de subunidades α, β, β’, σ, ω; existen dos copias de la subunidad α y una de cada una de las demás.
Cuando la subunidad σ se asocia al núcleo enzimático antes de su unión al ADN, se conforma una holoenzima capacitada para leer adecuadamente las secuencias promotoras. Por lo tanto, sigma se comporta como un factor de inicio de la transcripción, del cual depende la selectividad del proceso.
Los promotores procariontes poseen dos secuencias de bases conservadas o secuencias consenso que actúan como sitios de reconocimientoà para la unión de la holoenzima y señalización del punto de inicio; y funcionan como sitios de control de la expresión génica. Las secuencias consenso sonàTATAATo caja Pribnowen posición -10 y TTGACA en posición -35.
Cada factor σ transcribe determinados grupos de genes.
Cuando se inicia la transcripción, la holoenzima ARN polimerasa forma un complejo con el promotor. En principio es un complejo promotor cerrado, pero rápidamente la enzima cataliza es desenrollamiento del ADN, dando lugar al complejo promotor abierto. Cuando el transcripto añade alrededor de 8 nucleótidos, el factor σ se disocia de la ARN polimerasa y la transcripción es continuada por el núcleo de la enzima.
Las señales de terminación se dividen en dos clases: independientes de la proteína rho y dependientes de rho.
Terminación independiente de rho
Terminación dependiente de rho
TRANSCRIPCION EN EUCARIONTES
Es llevada a cabo por tres tipos de enzima ARN polimerasa, cada una especializada en la síntesis de distintos tipos de ARNs, y todas son proteínas cuaternarias constituidas por distintas subunidades.
|
Característica |
ARN polimerasa I |
ARN polimerasa II |
ARN polimerasa III |
|
Localización |
Nucléolo |
Nucleoplasma |
Nucleoplasma |
|
Productos transcriptos |
-ARN ribosómico grande (45 s). |
-Todos los ARNm -La mayoría de los ARN pequeños nucleares. |
-ARNt. -ARN ribosómico (5s). -ARN pequeño citoplasmático. -El resto de los ARN pequeños nucleares. |
|
Sensibilidada la α amanitina (toxina que afecta la actividad enzimática) |
No se ve afectada. |
Muy sensible. |
Moderadamente sensible. |
A diferencia de las procariotas, las enzimas eucariotas solo se unen al promotor por intermedio de proteínas llamadas factores basales de transcripción, cuya presencia garantiza el inicio de la transcripción.
Las células eucariotas también poseen factores de transcripción específicos, los cuales relacionan a los factores basales con las regiones reguladoras de un gen. Son proteínas reguladoras de genes que controlan la tasa de transcripción.
Los promotores para la ARN polimerasa II se ubican rio arriba del punto de inicio de la transcripción y comprenden tres sitios. Uno en la posición -25 es la caja TATA o caja de Hogness-Goldberg, secuencia consenso formada por restos de timina y adenina, ricas en secuencias GC, se encarga de alinear a la ARN polimerasa para que la transcripción se inicie en el sitio correcto. Otros sitios del promotor son las cajas CAATà posición -80, lleva secuencia GGCAATCT; y GCà ubicación variable, con secuencia GGGCCGGG.
La señal de terminación es desconocida. Sin embargo, la secuencia AAUAA, señal de poliadenilación, presente en los pre-ARNm (ARN transcripto primario) es reconocida por una endonucleasa que separa el producto de transcripción algunos nucleótidos rio debajo de la misma, poniendo fin al transcripto primario. La secuencia mencionada servirá para indicar el sitio correspondiente a la poliadenilación, un tercer proceso de modificación de los transcriptos.
No hay señal de poliadenilación en genes que codifican histonas. Y en algunos genes hay más de una señalà el mismo gen puede ser transcripto por dos productos diferentes, dependiendo de la señal reconocida.
Principales diferencias en la transcripción en células procariontes y eucariontes:
|
- Existe una sola ARN polimerasa procariota y tres ARN polimerasas eucariotas. - La ARN polimerasa procariota no requiere factores de transcripción. Las eucariotas requieren la presencia de factores de transcripción basales y su actividad es regulada por factores de transcripción específicos. - Las secuencias señalizadoras son distintas. |
EL CODIGO GENETICO
Muchos de los ARN transcriptos son productos celulares finales con una función propiaàla transcripción es un paso final de la expresión de ciertos genes. Pero muchos otros genes contienen la información necesaria paraespecificar la secuencia de aminoácidos de todas las proteínas que una célula puede sintetizar. Es estos casos, la transcripción es solo el primer paso de la expresión genética. Los ARNm obtenidos transportan información que debe traducirse en la síntesis de una proteína. La traducción es el segundo paso de la expresión genética.
La información que el ADNm lleva para construir una proteína reside en la secuencia de bases y esta “escrita” en un código genético.
Se puede pensar en el código genético como un idiomaà las letras son las 4 bases que forman las cadenas de ARN (A, C, U, G); las palabras son siempre agrupaciones de 3 letras o tripletes de bases, llamadas codones en la molécula de ARNm, y los objetos designados por dichas palabras son los 20 aminoácidos que componen las proteínas. Se obtienen 64 combinaciones diferentes si las bases se combinan de a 3, 64 codones son más que suficientes para nombrar los 20 aminoácidos.
El código genético emplea codones diferentes para nombrar a un mismo aminoácido. La mayoría de los aminoácidos están codificados por más de 1 codón: codones sinónimosà Su presencia habla de una degeneración que reporta a las células sus beneficios, y son muy similares entre sí.
El código genético no es ambiguoà cada codón especifica sólo a un aminoácidoà así no da lugar a error en el momento de ser traducido.
Los codones que codifican aminoácidos suman en total 61. Los 3 codones que no especifican ningún aminoácido, UGA, UAG y UAA, actúan como señales de terminación en la traducción o síntesis de proteínasàcodones determinación o stop.
El código genético es universalà todos los organismos comparten un mismo origen. A excepción de ADN mitocondrial, en el que algunos codones son leídos de manera diferente.
Marco de lectura
El sitio de inicio de la traducción es de gran importancia para que la traducción del mensaje sea correcta. Los ARNm portan un codón, el AUG (codifica metionina), que es interpretado por los ribosomas como codón de iniciación. Este codón da el marco de lectura que será empleado de allí en más. En adelante, el ribosoma se desplaza a lo largo del ARNm en bloques consecutivos de tres bases, garantizando la lectura de los codones apropiados.
Mutaciones:
No se coloquen todas las bases.
Se coloque una base de más.
Sustitución de bases.
Resultado:
Va a haber un producto final diferente al que se espera.
Dura en el ARNm hasta su degradación.
Durante la transcripción.
Las mutaciones que implican la ganancia o pérdida de un nucleótido son más destructivas que las sustituciones, ya que al modificar el marco de lectura, alteran por completo el mensaje.
El código es leído sin solapamientoà cada nucleótido del mensaje es usado como componente de un solo codón.
Características del código genético:
|
- Es un conjunto de todos los tripletes de bases y sus significados. - Consta de 64 codones o tripletes de bases. - 61 codones codifican aminoácidos. - 3 codones funcionan como señales de terminación. - No es ambiguoà cada codón especifica un solo aminoácido. - Es degeneradoà un aminoácido puede estar codificado por diferentes codones. - Es universalà sus mensajes son interpretados de la misma forma por todos los organismos. - Utiliza un marco de lectura establecido al inicio de la traducción y no lo modifica. - No se produce solapamiento de codones. - Lectura en sentido 5’=>3’ |
MAQUINARIA TRADUCCIONAL
La traducción implica el funcionamiento coordinado de un gran número de moléculas entre las que se encuentran los distintos tipos de ARN. Una vez transcriptos cada ARN sufre modificaciones que varían según sean procariotas o eucariotas.
ARNm
EUCARIOTA
1- Secuencia del mensaje interrumpidaà coexisten a lo largo de la molécula à EXONES: sectores que codifican para la proteína, e INTRONES: secuencias intercaladas sin información.
2- Los ARNm transcriptos primarios sufren una serie de modificaciones post-transcripción antes de salir como ARNm maduro.
Capping: se adiciona en el extremo 5’ del ARNm una molécula denominada cap. Es una modificación co-transcripcional. El cap impide la degradación del ARNm inmaduro por nucleasas y fosfatasas nucleares. También participa en remoción de intrones e inicio de la traducción.
Poliadenilación: agregado de 250 adenosinas o cola poli A, en el extremo 3’ del ARNm. Una nucleasa corta al pre-ARNm a unos 20 nucleótidos de la señal de poliadenilación. Una vez liberado el pre-ARNm la poliA-polimerasa le agrega las adenosinas de a una por vez. Las funciones de la cola poli A son: proteger el extremo 3’ de la degradación y ayudar a los ARNm a salir del núcleo.
Otra modificación es causada por el acortamiento de la secuencia, producto de la eliminación de los intrones quedando como producto final los exones empalmados en secuencia continuaàprocesamiento empalme (splicing)à es un tipo de maduración del pre-ARNm y para que se lleve a cabo necesita de las ribonucleoproteinas nucleares RNPpnà U1, U2, U4, U5, U6, se combinan en los extremos de cada intron. El complejo resultante del ensamblado es el espliceosoma. Los ARNpn del espliceosoma se encargan de reconocer las secuencias señalizadoras de corte, separar los intrones y empalmar a los exones entre ellosà produciendo una molécula de ARNm maduro.
3- Los ARNm maduros son monocistrónicosàel sector codificador dicta la secuencia para una sola cadena polipeptidica.
PROCARIOTA
1- Los ARNm procariotas presentan las secuencias codificadoras continuasà carecen de intrones.
2- No sufren modificaciones post-transcripcionales.
3- Muchos ARNm procariotas son policistrónicosà una sola molécula de ARNm contiene información para varias proteínas. Este ARNm se traduce como varias moléculas de proteínas distintas.
ARNt
Son moléculas adaptadorasà interactúan por un lado con la cadena polinucleotídica(ARNm) y por el otro con los aminoácidos que formaran parte de la cadena polipeptídica, asi alinean a los aminoácidos siguiendo el orden de los codones del ARNm.
Enlaces puentes de hidrogeno entre las bases repliegan la molécula y le dan forma de trébol.
Se distinguen dos extremos:
El extremo 3’àextremo aceptor, en el que se encuentra el trinucleótido CCAà sitio de unión donde se liga el aminoácidoà la unión es catalizada por una enzima aminoacil ARNt sintetasaespecifica y apropiada para cada uno de los 20 aminoácidos.
En el otro extremo de la Là un trinucleótido, anticodónà su secuencia varía según el tipo de ARNtà cada uno es complementario de un codón de ARNm.
Las modificaciones de los pre-ARNt son parecidas en procariotas y eucariotas, en cuanto en ambos se producen modificaciones químicas.
Propiedades especificas:
Debe ser reconocido por un aminoacil ARNt sintetasa que lo una al aminoácido correcto.
Debe tener una región que actué como sitio de unión para el aminoácido.
Debe tener una secuencia complementaria (anticodón) específica para el codón del ARNm correcto.
ARNr
Junto con las proteínas, son los componentes de los RIBOSOMAS. Intervienen en la traducción de la información codificada en el ARNm.
Cada ribosoma tiene dos subunidadesà la subunidad MAYOR y la subunidad MENOR. El ARNm se inserta en el surco que se forma entre las subunidades. Las subunidades trabajan en conjunto para la síntesis de proteínas.
Dentro del ribosoma hay dos huecos llamados sitio A (AMINOACÍDICO) y sitio P (PEPTIDÍLICO) para el ingreso de los ARNt unidos a sus aminoácidos correspondientes.
Los ribosomas procariotas y eucariotas cumplen la misma función, pero se diferencian en su tamaño, coeficiente de sedimentación, y en el tipo y numero de moléculas de ARNr y proteínas que los forman.
Todos los ARNr procariotas y eucariotas sufren modificaciones post-transcripción.
Las unidades ribosómicas por separado y antes de completar sus ensambles son exportadas al citoplasma a través del complejo del poro, concluyendo el procesamiento de casa subunidad en el citosol.
ARN PEQUEÑOS
Forman complejos con proteínas específicas formando partículas ribonucleoproteicas (RNP):
Las RNP del núcleo se llaman RNPpnàvarias participan en el procesamiento de los ARNmà forman un complejo multienzimático, espliceosomaà encargado de realizar cortes y empalmes en los ARNm transcritos primarios.
Las RNP del citoplasma se denominan RNPpcàsu función más conocida la desempeña el complejo ARN/proteínasà componen la partícula de reconocimiento de la señal (SRP).
EL PROCESO DE LA TRADUCCION O SINTESIS PROTEICA
La síntesis proteica consiste en la traducción de la información codificada en la secuencia de nucleótidos del ARNm, en la secuencia correspondiente de aminoácidos en una cadena polipeptídica.
La síntesis hace en los ribosomas, y la maquinaria traduccional es más compleja en eucariotas.
La síntesis proteica incluye dos etapas:
1-Activación de los aminoácidos o aminoacilación.
Antes de la traducción cada ARNt se engancha a su aminoácido específico. Esta reacción de aminoacilación es catalizada por las enzimas aminoacil ARNt sintetasas, y existe una distinta para cada aminoácido.
Este proceso ocurre en dos pasos:
a- Se usa la energía de la hidrólisis de ATP para unir cada aminoácido a un AMP con un enlace de alta energía. Esta reacción de origen a un complejo intermediarioàaminoacil-AMP.
b- Sin abandonar la enzima, se lleva el aminoácido del complejo aminoacil-AMP al ARNt especificoàse origina la molécula finalàAMINOACIL-ARNtàpresenta dos sitios activos, uno para la unión de ARNt y otro para su aminoácido especifico. Una vez acoplado el aminoácido a su ARNtàel complejo aminoacil-ARNt se une a un codón del ARNm. De esta manera, el ARNt determina el lugar en el que será añadido el aminoácido durante la síntesis proteica.
2- Traducción del ARNm.
Es el mecanismo por el cual se traduce el mensaje del ARNmà se describe en tres etapas:
Iniciaciónàfactores de iniciación. Complejos sistemas de proteínas.
Elongaciónàfactores de elongación. Son diferentes en procariotas y eucariotas,
Terminación àfactores de terminación. aunque sus funciones son parecidas.
INICIACION DE LA TRADUCCION
Se reúnen los componentes que forman el complejo de iniciaciónàdisparador de la síntesis proteica. Formado por un ARNm, una subunidad mayor, una subunidad menor, el ARNt iniciador y factores proteicos de iniciación.
Posible secuencia:
1- El aminoacil-ARNt iniciador se una a la subunidad menor, con gasto de GTP.
2- El ARNm se acopla a la subunidad menor.
3- La subunidad menor se desliza sobre el ARNm hasta encontrar el codón AUG. Este modelo de selección de eucariotas difiere del modelo de emparejamiento de procariotas, en el que se emparejan AUG del ARNm con el extremo 3’ del ARNr de la subunidad menor.
4- Una vez encontrado y acoplado el anticodón al codón AUG del mensaje se establece la pauta de lectura correcta para el resto de los codones que contenga la región codificadora del ARNm.
5- Finalmente, los factores de iniciación se acoplan a la subunidad mayor, quedando el aminoacil-ARNt iniciador en el sitio P del ribosoma.
|
Tres clases de interacciones determinan el inicio de la síntesis proteica: - Reconocimiento del codón de iniciación AUG en la subunidad menor del ribosoma. - Acoplamiento de bases del codón AUG con el ARNt iniciador. - Acoplamiento de la subunidad mayor del ribosoma cerrando el complejo de iniciación. |
ELONGACION DE LA CADENA POLIPEPTIDICA
Se puede resumir en tres etapas:
6- Una molecula de aminoacil-ARNt entra al sitio A del ribosoma acoplándose al segundo codón del ARNm expuesto en ese lugar.
7- El aminoácido iniciador se desacopla del ARNt del sitio P, liberando energía que se usa en la formación del enlace pepitídico entre los dos aminoácidos alineados. Esta reacción es catalizada por una peptidil transferasa integrante de la subunidad mayor. Como consecuenciaà el ARNt iniciador del sitio P queda sin aminoácido y el dipéptido resultante queda enganchado al ARNt del sitio A (peptidil ARNt).
8- El nuevo peptidil ARNt del sitio A es movido al sitio P cuando el ribosoma se desplaza tres nucleótidos a lo largo del ARNm.
|
El ciclo de elongación se caracteriza por: - La unión aminoacil-ARNt (reconocido por el codón). - La formación del enlace peptidico. - La translocación del ribosoma. |
TERMINACION DE LA SINTESIS PROTEICA
9- Ocurre cuando uno de los tres codones de stop llega al sitio A del ribosoma.
10- Como consecuencia de esta reacción el polipéptido se desacopla del ARNt, liberándose en el citoplasma. El ARNm se desacopla del ribosoma y se disocian las dos subunidades, las cuales podrán volverse a ensamblar sobre otra molécula de ARNm reiniciándose el proceso de traducción.
|
- Reconocimiento del codón de stop. - La disociación de las subunidades ribosómicas, el ARNm y la cadena polipeptídica. |
POLIRRIBOSOMAS
Al grupo de ribosomas que traducen simultáneamente al mismo mensaje se lo denomina polirribosoma. Los ribosomas en esta unidad estructural trabajan independientemente sintetizando una cadena polipeptídica completa.
DIFERENCIAS EN LA TRADUCCION
En eucariotas el ARNm es “leído” después de que haya abandonado el núcleo a través de los poros nuclearesà la traducción es post-transcripcional.
En procariotas la traducción es simultánea a la transcripciónà mientras se está terminando de transcribir el extremo 3’, el extremo 5’ libre del ARNm se asocia a un ribosoma y al ARNt iniciador comenzando la traducción.
REGULACION DE LA EXPRESION GENTICA
REGULACION EN PROCARIOTAS: EL OPERÓN
En procariotas se puede regular de varias formas la cantidad de proteínas que van a ser sintetizadas, aunque se considera que la expresión génica está regulada principalmente a nivel de la transcripción.
Los genes que codifican para la síntesis de enzimas que participan en una vía metabólica se agrupan en el cromosoma en un conjunto denominado OPERÓNàTodos sus genes actúan como unidades coordinadas mediante un mecanismo de control.
Ese mecanismo de control consta de:
Genes estructuralesàson los genes que codifican las enzimas relacionadas o proteínas estructurales. Están cerca entre sí y por eso son transcriptos por un mismo ARNm policitrónicoà y cuando este se traduce se obtienen distintas enzimas de la vía metabólica.
Promotorà secuencia de nucleótidos del ADN en donde se une la ARN polimerasa para empezar la transcripción.
Operadorà secuencia de nucleótidos que se interpone entre el promotor y los genes estructurales, es donde se inserta una proteína reguladora llamada proteína represora, obstruyendo al promotor del gen estructural. El operador controla el acceso de la ARN polimerasa al promotor. Cuando la proteina represora es removida se puede iniciar la transcripción.
La proteína represora es codificada por el gen reguladorà controla el tiempo y velocidad de la transcripción de otros genes. Y se encuentra en otra parte del cromosoma, antes del sitio operador.
El ejemplo de operón más conocido esàoperón lacàconjunto de genes que intervienen en el uso de lactosa como fuente de energía. Las tres enzimas que intervienen en la degradación de la lactosa son: la permeasa, la beta galactosidasa y la transacetilasa.
El operón lac está formado por tres genes estructurales en serieàlacz, lacy, lacaà su transcripción da origen a un ARNm que codifica para las tres enzimas.
En ausencia de lactosaà el represor se enlaza al operador e impide a la ARN polimerasa insertarse en el promotorà se interrumpe la transcripción. La influencia del represor es mediante control negativoà su interaccion con el ADN inhibe la expresión del operón.
En presencia de lactosa à la lactosa se une al represor y lo inhibeà se transcriben los genes estructurales, apareciendo en el citosol las enzimas que degradan la lactosa.
El operón lac es un operón inducibleàla presencia de una sustancia especifica (lactosa) produce la transcripción de los genes estructurales.
El operón lac está bajo control positivoà cuando en el medio hay glucosa. Cuanto menor es la concentración de glucosa en el medio, mayor es la concentración de AMPcàtiene influencia en el “encendido” del operonlacà cuando su concentración es alta se fija a un sitio del promotor lac, aumentando su afinidad por la ARN polimerasa.
|
Para que se exprese el operonlac deben darse dos condiciones en el medio: presencia de lactosa y baja concentración de glucosa. |
Operón reprimibleà cuando un producto del metabolismo esta en cantidades suficientes la bacteria puede dejar de fabricar las enzimas que lo sintetizan.
Dentro de los operones reprimibles, está el operón triptófano(producto)à 5 genes estructurales que codifican las enzimas involucradas en la biosíntesis del aminoácido triptófano. El triptófano funciona como correpresor uniéndose al represor y de ese modo detiene la síntesis proteica.
En ausencia de triptófano, la ARN polimerasa se une al promotor y transcribe los genes estructurales en un ARNm policistrónicoà ya que el represor inactivo no logra unirse por sí solo al operador.
En presencia del triptófanoà este se une al represor formando el complejo co-represorà se fija al operador e impide que la ARN polimerasa transcriba los genes estructurales.
|
OPERÓN LAC |
OPERÓN TRIPTÓFANO |
|
Operón inducible, se expresa en presencia de lactosa. |
Operón reprimible, se expresa en ausencia de triptófano |
|
La lactosa es el inductor. |
El triptófano es el co-represor. |
|
El represor se sintetiza en forma activa. Actúa solo. |
El represor se sintetiza en forma inactiva. Actúa en presencia del co-represor. |
|
Sus enzimas participan en una vía catabólica. |
Sus enzimas participan en una vía anabólica. |
REGULACION EN EUCARIOTAS
Los distintos tipos celulares se diferencian entre sí porque fabrican y acumulan distintos ARNm y por lo tanto distintas proteínas.
Cada etapa en el flujo de información “ADNàARNàPROTEINAS” puede ser regulada.
Algunos mecanismos de regulación en eucariotas:
Mecanismos de control a nivel transcripcional
- A) Los factores de transcripción y la expresión genética
Para la transcripción de un gen eucariota se requiere:
Promotorà secuencia de nucleótidos necesaria para la fijación de la ARN polimerasa.
Secuencias reguladorasàIntensificadoras: estimulan la transcripción y pueden estar muy lejos del promotor “rio arriba” o “rio abajo”
àSilenciadoras: inhiben la transcripción. También muy distantes del promotor.
No existen dos genes que tengan la misma combinación de estas secuencias.
Factores basales de transcripciónà complejo proteico que interacciona con el promotor. Esenciales para la transcripción.
Factores específicos de transcripciónà proteínas reguladoras que pueden ser activadoras o represoras.
àActivadoras: interaccionan con las secuencias intensificadoras del gen.
àRepresoras: interaccionan con las secuencias silenciadoras del gen.
B)La estructura de la cromatina y la expresión genética
El grado de compactación de la cromatina influye mucho en la expresión génica.
Eucromatinaàtranscripcionalmente activa y mas desplegada.
Heterocromatinaàtranscripcionalmente inactiva y mas compactada.
La transcripción solo ocurre cuando el ADN esta mas desplegado.
- C) Grado de metilación y la expresión genética
La mayoría de los genes que no se expresan están metilados, mientras que en otra celula donde esos genes se expresan se nota una disminución de sus niveles de metilación.
Mecanismos de control a nivel procesamiento del ARNm
Empalme alternativoà mecanismo por el cual puede obtenerse de un mismo gen dos proteínas relacionadas.
Mecanismos de control a nivel de traducción
Cuando la concentración intracelular de hierro es baja, la aconitasa (proteina represora) se une a una secuencia nucleotídica especifica del ARNm bloqueando la traducción.
Mecanismos de control después de la traducción.
Modificación de la proteína; control del plegamiento; vida media de las proteínas.
CICLO CELULAR
Las nuevas células se originan a partir de otras células por medio de la división celular. Las células hijas heredaran la misma información genética que poseía su progenitora, la célula madre. Al llegar a la madures las células hijas se dividirán, transmitiendo los mismos genes que heredaron a sus propias células hijas. Por lo tanto la división celular constituye el enlace entre un progenitor y su descendencia. La célula sufre una línea temporal continua, se habla de ciclo porque en cada nueva generación se van a suceder los mismos procesos.
Las etapas por las que pasa la célula desde una división celular a la siguiente constituyen el ciclo celularà se divide en dos fases principalesà la fase M (mitótica) y la intrafase.
El proceso de la división celular (fase M) consiste en dos procesos secuenciales: la división nuclear (cariocinesis-mitosis) y la división citoplasmática (citocinesis). Pero antes de que una célula pueda dividirse, debe duplicar su masa y todos los elementos que contiene. Solo así es posible que las dos células hijas contengan todos los componentes que necesitan para iniciar su propio ciclo.
La mayor parte del trabajo necesario para la preparación de la división celular se produce durante la fase de crecimiento del ciclo celularà interfase. En la síntesis de ADN, esta fase se divide en tres etapas:
Fase G1à es la fase de crecimiento de la célula, duplica su masaà síntesis activa de proteínas y ARN, y duplicación de todas sus organelas.
Fase Sàreplicación del material genéticoà se duplicación y síntesis del ADN, síntesis de histonas (necesarias) y de los ARNm específicos para estas.
Fase G2à es la última fase, durante la cual la célula finaliza su crecimiento y se prepara para la divisiónà realiza la síntesis los elementos requeridos para la mitosis.
Sin considerar los casos extremos, el ciclo de las células somáticas animales se repite aprox. cada 18-24 horas
G1 S G2 M
|
0hs |
2hs |
4hs |
6hs |
8hs |
10hs |
12hs |
14hs |
16hs |
18hs |
20hs |
22hs |
24hs |
Variaciones en la duración del ciclo celular
Existen por lo menos tres tipos de células con ciclos celulares “atípicos”:
- Algunas células con especialización estructural extrema no se dividenà permanecen en estado àsemejante a G1, pero son incapaces de proseguir a la fase S. Por ejemplo, células nerviosas y muscularesà neuronas, glóbulos rojos.
- Células que normalmente no se dividen, pueden abandonar G0 y reingresar al ciclo cuando reciben un estímulo apropiado. Por ejemplo, células hepáticas y linfocitos.
- Células con alto nivel de actividad mitóticaà ciertos tejidos sujetos a renovación continua y deben formarse permanentemente nuevas células mediante división celular. Ejemplo: células germinales (ovogonias y espermatogonias) que forman las gametas, células epiteliales.
CONTROL DEL CICLO CELULAR
Mecanismo de regulación para que la división celular se de en el orden correcto. El paso de una célula eucariota por el ciclo celular requiere de la integración de varias señales intra y extra celulares; si una de las señales no está presente la célula fallara en el paso de una fase del ciclo a la otra.
Factores promotores de las transiciones del ciclo celular
Los distintos reguladores del ciclo celular se descubrieron a partir de la fusión de células en distintas fases del ciclo. La fusión celular se desarrolla en presencia de agente que causan la unión de las membranas plasmáticas formando una célula hibrida llamada heterocarion(dos o más núcleos dentro de un citoplasma rodeado por una sola membrana plasmática).
|
|
|
HETEROCARION |
|
|
CELULA 1 X CELULA 2 |
NUCLEO 1 |
NUCLEO 2 |
FACTOR |
|
Fase S x Fase G1 Fase S x Fase G2 Mitosis x Interfase Fase G1 x Fase G2 |
Replicación Replicación Mitosis Interfase |
Replicación Demorado CPC (prematura condensación de los cromosomas) Interfase |
FPS FPS (?) FPM |
Las células en fase S contienen un factor promotor de fase S capaz de inducir una fase S prematura en las células en fase G1, peo no en fase G2. Por otro lado, las células en fase M contienen un factor promotor de fase M capaz de inducir eventos mitóticos en células en cualquier estadio del ciclo celular.
FPS y FMS son quinasas dependientes de ciclinas (CDKs)à complejo de enzimas, en su forma activa, compuesto por una subunidad catalítica, la quinasa y una subunidad regulatoria, la ciclina.
Las quinasas son enzimas que catalizan la fosforilación de proteínas que permiten que se lleve a cabo la fase S o Mà puede activar o desactivar la proteína modificada, según el caso. Su concentración es constante a lo largo del ciclo.
Las ciclinas regulan la actividad de la quinasa de las CDKs:
à cuando la concentración de ciclina es baja, la quinasa es inactiva.
àcuando la concentración de la ciclina es suficiente, la quinasa se activa y desencadena la entrada de la célula a la fase siguiente correspondiente.
Su expresión es regulada, ya que su concentración varia en forma cíclica a lo largo del cicloà crece (se sintetizan) y decrece (son degradadas)
FPSà formada por CDK y la ciclina G1.
àFosforila proteínas claves para el origen de la replicación. Una serie de bases dan la señal para el inicio de la duplicaciónà esta secuencia está unida al CRO (complejo de reconocimiento de la replicación).
En G1à el CRO interacciona con las proteínas A y B, que forman el complejo pre-replicativoà indica que se puede iniciar la replicación del ADN.
FPMà formado por CDK y la ciclina mitótica (M)
àCataliza la fosforilación de la lámina nuclear, provocando la ruptura de la envoltura nuclear.
àCataliza la fosforilación de histonas, provocando la condensación de los cromosomas.
àFragmentación del Golgi y el RE.
àResponsable de la fragmentación del Huso mitótico y de la alineación de los cromosomas en la placa metafísica.
Salida de la fase Mà destrucción de la ciclina mitótica, por ende, desaparece el complejo CDK-ciclina M.
Las CDKs requieren niveles adicionales de regulación que dependen de:
Fosforilación y desfosforilación de la subunidad catalítica de la CDK.
Presencia de proteínas inhibidoras capaces de interactuar con las CDKs (CDKI).
Proteólisis(degradación de proteínas)controlada por las ciclinas.
Puntos de control o checkpoints
Controla los puntos clave del ciclo celular y solo lo deja avanzar si estos han sido completados.
Son vías inhibitorias, aseguran la dependencia de un proceso del otro.
Punto de control del ADN no replicadoà si una célula no duplica correctamente sus cromosomas, NO entra en mitosis. Es medido por proteínasà una reconoce la fase de replicación y activa su actividad quinasaà el complejo CDK-ciclina unido al inhibidor no da comienzo a la mitosis.
Punto de control del ensamblado del husoàchequea que los cinetocoros se hayan unido correctamente a los microtúbulos del huso. La proteína detecta cinetocoros libres y bloquea el inicio de la anafase.
Punto de control de la segregación de los cromosomasà los cromosomas hermanos no segregan correctamenteà no se inicia la telofase.
Punto de control del ADN dañadoàArresto celularà bloque el ciclo celular hasta que se repare el daño. Se evita la copia de secuencias de nucleótidos dañada. Daño del ADNà activación de una proteína quinasaàdos vías de respuestasà activa las fosfatasas que inhiben las CDKs.
àPresencia de sities genotóxicos (causan daño al ADN).
Aumenta la concentración de la proteína P53 (activador de genes). El aumento es transitorio, si se mantienen los daños aumenta por más tiempo.
P53à inhibe CDKs deteniendo la interfase.
Inhibe proteínas esenciales para la replicación del ADN.
Cáncerà enfermedad producida por alteraciones genéticas vinculadas con el control celular, resultando en la formación de tumores invasivos que pueden propagarse a distancia por la liberación de células.
REPLICACION DEL ADN
Síntesis del ADNà ocurre en la fase S.
Es una autoduplcaciónà el ADN lo hace solo.
Proceso metabólico de tipo anabólicoà exergónico. Los sustratos son los desoxirribonucleótidos trifosfatados y el producto la molécula de ADN.
La energía la aportan los mismos sustratos de sus enlaces 1° fosfato y 2° fosfato. Igual que en la transcripción
Se necesitan enzimas ADN-POLIMERASA, hay distintos tipos: helicasa, topoisomerasa, primasa, ADN-ligasa.
El molde es la propia molécula de ADN.
Propiedades universales de la replicación
La replicación del ADN es semiconservativa
Tres posibles mecanismos de replicaciónà 1) Conservativo, en el cual la replicación producía una molécula hija de ADN completamente nueva y otra que conservaba las dos cadenas originales. 2) Semicoservativo, en el cual se producían dos moléculas hijas formadas por una cadena original y otra nueva. 3) Dispersivo, ambas cadenas de las dos moléculas producidas estaban compuestas por fragmentos nuevos y fragmentos originales.
La replicación tiene un sitio de origen y ocurre en forma bidireccional
La replicación tiene sitios específicos en los que se generan las horquillas de replicación (forma de Y) determinando un proceso bidireccional. Las horquillas representan el sitio donde la doble hélice parental rompe sus puentes de H permitiendo la entrada del aparato enzimático que empezara la polimerización de las cadenas hijas utilizando como molde las cadenas parentales.
La replicación es antiparalela y la síntesis de ADN es siempre en sentido 5’à 3’
La ADN-polimerasa tiene 5’à 3’ como único sentido de síntesis. Solo una de las dos cadenas se sintetiza en forma continua (cadena adelantada), utilizando como molde la hebra parental 3’à 5’ (mismo sentido que la horquilla). La otra cadena hija (cadena retrasada) debe ser sintetizada necesariamente en forma de pequeños trozos denominados fragmentos de Okazaki, que crecen en sentido contrario a la horquilla.
Proceso de duplicación
INICIACION: la unión de la proteína de iniciación al sitio de origen da comienzo a la formación de la horquilla de replicaciónrompiendo las uniones puente de H entre las bases complementariasà las HELICASAS ingresan al sitio creando dos horquillas de replicación, c/u se desplaza en sentido opuesto (bidireccionalidad). A ambas cadenas simples se unen proteínas desestabilizadoras (las SSDPà proteínas de unión de la cadena simple de ADN) que las mantienen separadas impidiendo que se vuelvan a juntar.
ELONGACION: la helicasa rompe los puentes de hidrogeno permitiendo el avance de las horquillas de replicación y la enzima GIRASA (topoisomeasa) aliviala tensión molecular delate de la horquillapor esta aperturaà la girasa corta las cadenas de ADN y luego la ligasa las vuelve a juntar.
La ADN-POLIMERASA III es la encargada de producir las cadenas nuevasà trabaja con desoxirribonucleótidos en sentido 5’à 3’. Esta agrega desoxirribonucleótidos al extremo 3’ OH generando un enlace fosfodiéster. Pero el extremo 3’ no está libre y necesita algo que lo libere, sino esta no puede comenzar colocando el primer nucleótido.
La enzima ARN-PRIMASA produce una corta cadena de ARN antiparalela que le va a dar a la ADN-pol III el extremo 3’ libre que necesita para trabajar. Este se llama PRIMER. Una vez formado el primer, la ADN-pol III lee la cadena molde y une los nucleótidos por complementariedad de bases, siendo la cadena nueva antiparalela a la cadena molde.
La ARN-polimerasa I elimina los primers y los reemplaza por fragmentos de ADN. La ADN-ligasa une los fragmentos de Okazaki entre sí.
Hay mecanismos que corrigen erroresà la ADN-polimerasa III es capaz de retroceder sobre sus pasos, revisarlos y reportar un problemaà CORRECCION DE PRUEBA.
La ADN-polimerasa II se encarga de reportar mutaciones.
TERMINACION: comprende el encuentro de ambas horquillas de replicación en un punto opuesto al sitio de origenà da como resultado la separación de las dos moléculas de ADN.
Similitudes y diferencias en la síntesis de ADN entre procariontes y eucariontes
Comienzan en orígenes de replicación que es una secuencia del ADN que indica que ahí hay que empezar.
En el procarionte hay un único origen de replicación.
Los eucariotas tienen varios orígenes de replicación.
Le velocidad de movimiento de la horquilla de la replicación en eucariontes es mucho más lenta porque hay mucho más ADN y más concentrado. En los procariontes es más rápido, pero tiene un solo origen.
En eucariotas, exclusivamente, ocurre el ACORTAMIENTO DE TELOMEROSà cuando se elimina el primer, no es completado el fragmento y queda vacío. Y lo mismo pasa en la otra cadena y en el otro extremo.
En las células donde no ocurre esto hay una enzima llamada TELOMERASA, que alarga la cadena molde de modo que haya más espacio y construir un primer nuevo.
DIVISION CELULAR
EN PROCARIOTASà reproducción asexualàun solo individuo, no hay gametas y no genera variabilidad genética. Por fisión binariaàreplicación del cromosomaà se alarga el citoplasmaà nacen dos células hijasà cada celula hija adquiere un cromosoma que ya ha comenzado a replicarse nuevamente.
Los individuos intercambian material genético por los mecanismos de transformación, transducción, y conjugación.
EN EUCARIOTASàMitosisàes un tipo de división celular mediante la cual a partir de una célula madre se obtienen dos células hijas idénticas entre sí, cada una con la misma información genética y por lo tanto con la misma cantidad de cromosomas.
Consta en dos subetapas:
Cariocinesisàabarca la división del material nuclear para la formación de los núcleos hijos.
Citocinesisà es la separación del citoplasma para dar origen a las células hijas.
Etapas de la mitosis
La mitosis es un proceso continuo, sin etapas definidas, pero se identifican cinco fases dentro de la CARIOCINESIS:
ProfaseàEs la etapa más larga de la mitosis y se divide enà profase temprana y profase madura.
Profase tempranaà empieza a desarmarse la envoltura nuclearà para esto tiene que desarmarse la lámina nuclearà esto lo hacen los FMP. Y empieza a formarse el huso de división.
Condensación de la cromatinaàel nucléolo desapareceà lo que antes eran largas moléculas de ADN ahora se compactan y forman una parte del cromosoma (2 moléculas de ADN condensadasà cromátidas hermanasàson idénticas)
Fuera del núcleoà los centriolos se duplican y empiezan a separarse formando una estructura de microtúbulos.
Profase tardíaà quedan vesículas pequeñas (fragmentos de la envoltura nuclear). El Huso de división bien formado ocupando todo el centro de la célula y los cromosomas en el plano ecuatorial.
Ploidaà numero de juegos cromosómicos que tiene una célula.
Diploideà 2 juegos cromosómicosà 2n=46 (humanos).
Cromosomas homólogosà igual forma y tamaño.
Metafaseà cromosomas alcanzan su máximo estado de condensación y son alineados en el plano ecuatorialà la molécula esta lista para ser separada. Si los cromosomas no están bien alineadosàmutación.
Anafaseà separación de las cromatides hermanas. La célula se alarga gracias a los microtúbulos polares. De esta forma se duplica el material genético de la célula. Las cromatinas que en un principio se encuentran en el plano ecuatorial, migran hacia los polos de la célula. Y el material genético para este momento ya ha sido duplicado.
Telofaseà se puede considerar inversa a la profaseà empiezan a descondensarse los cromosomas y vuelven al estado de la cromatina. Empieza a reorganizarse el núcleoà vuelve el nucléolo. Se forman dos nucleos independientes y surgen dos células hijas separadas.
CITOCINESIS
Es la equitativa partición y separación del citoplasma entre las dos células hijas para completar la mitosis. Generalmente comienza durante la telofase.
En eucariotas animalesà ocurre por estrangulamiento del citoplasma debida a la acción de un anillo contráctil en el plano medio de la célula que se va a dividir. El anillo está constituido por filamentos, está unido a la cara citoplasmática de la membrana mediante proteínas de anclaje y provee la fuerza necesaria para la contracción del citoplasma. La célula se profundiza progresivamente reduciendo el contacto entre las células hijas hasta la separación total de estas.
En plantasà se da por tabicamiento debido a que poseen una pared celular rígida que impide su estrangulación. El citoplasma se divide por la formación de membranas y una pared en el plano ecuatorial de la celula madre, separándola en dos compartimientos que serán las células hijas. La unión de las membranas de las vesículas dan por resultado la placa celular.
Funciones:
Reproducción asexualà transmite la información genética exacta de padres a hijos.
Indispensable para la cicatrización de los tejidos dañadosà las células entran en mitosis hasta cubrirla.
Crecimiento de todos los organismos pluricelulares.
Origen de nuevas plantas.
MEIOSIS Y REPRODUCCION SEXUAL
La reproducción es el mecanismo que permite generar individuos de la misma especie, preservando el número cromosómico a través de las sucesivas generaciones, y garantizando su perdurabilidad.
Participan dos individuos en la mayoría de los casos; 1 individuo en caso de hermafroditismo o partenogénesis.
El nuevo individuo surge como consecuencia de la unión de dos células sexuales o gametas, por el proceso de fecundación, originando la célula huevo o cigota. Las gametas femeninas y masculinas se forman en órganos especializados. En la fecundaciónà surge una única célula huevo o cigota a partir del ovulo y el espermatozoide.
En humanos el genoma está compuesto por 46 cromosomas, cada gameta aporta un juego completo de 23 cromosomas, llamado complemento haploide o “n”. La cigota resultante porta dos juegos completos de cromosomas llamados en conjunto complemento diploide o “2n”, donde los cromosomas concurren de a pares denominados “pares de homólogos”. Los integrantes de cada par son idénticos en forma y en tamaño y, llevan los mismos genes, aunque no necesariamente los mismos alelos para cada uno de ellos. La ventaja evolutiva de la reproducción sexual es que genera variabilidad genética.
La meiosis es un tipo de división celular que ocurre por única vez en células especializadas o germinales (2n) para dar cuatro células hijas haploides o gametas con nuevas combinaciones genéticas. Consiste en dos divisiones nucleares sucesivas, conocidas como meiosis I y meiosis II, precedidas por una única duplicación del ADN. La meiosis I es reduccionalà separa los miembros de cada par de homólogos entre sí. La meiosis II es ecuacionalà separa las cromátides hermanas de cada cromosoma.
Metafase I:
Profase Ià de una celula 2n=4à
Desintegración de la envoltura nuclear.
Formación del huso de división.
Los cromosomas se aparean por sinapsis originando bivalentes o tétradas.
ENTRECRUZAMIENTO o crossing-overà mecanismo de recombinación homologa entre ambos cromosomas (provenientes de individuos distintos)à surgen cromátides recombinantes y no recombinantes.
Desinapsisà los bivalentes se unen a las fibras del huso y comienzan a desplazarse hacia la placa ecuatorial, desaparecen los nucléolos y se produce la ruptura de la membrana nuclear.
Metafase Ià cromosomas homólogos apareados alineados en el plano ecuatorial.
Anafase Ià separación al azar de los cromosomas homólogos y su migración hacia los polos de la célula.
Telofase Ià reconstrucción nuclear una vez que los cromosomas llegan a los polos.
Cada nuevo núcleo es haploide aunque sus cromosomas todavía no se encuentren duplicados.
Según la especie puede o no ocurrir citocinesis.
Profase IIà los cromosomas vuelven a condensarse, se disgrega la membrana nuclear, desaparece el nucléolo y empiezan a aparecer nuevas fibras del huso.
Metafase IIà los cromosomas se ubican en el plano ecuatorial y, a diferencia de la metafase I, los cinetocoros de cromátides hermanas se enfrentan a polos opuestos.
Anafase IIà las cromátidas hermanas se separan y migran hacia polos opuestos.
Telofase IIà los husos desaparecen, se forma la envoltura nuclear en torno de cada juego de cromosomas. Simultáneamenteàcitocinesis dando 4 células haploides como resultado.
Gametogénesis
Es el proceso de formación de las gametas o células sexuales y ocurre en la meiosis terminal o gamética. En vertebrados, comprende la ovogénesis (formación del ovulo) y la espermatogénesis (formación del espermatozoide), procesos que tienen lugar en las gónadas u órganos sexuales femeninos y masculinos respectivamente.
Ovogénesis
Ocurre en los ovarios.
Células primordialesà ovogonias (2n).
En el tercer mes de desarrollo fetalà todas las ovogonias se dividen repetidamente por mitosis. Y cuando el número de ovogonias es el adecuadoà duplican su ADN (cromosomas dobles) y se diferencian (expresan determinados genes) originando ovocitos primarios u ovocitos I (2n).
Al quinto mes de vida intrauterinaà el ovocito I comienza la meiosis I.
En el octavo mes se detiene en profase I por años, hasta que la mujer madura sexualmente.
Durante la profase I los ovocitos I sintetizan una cubierta (que los protege de daños mecánicos), gránulos corticales (que durante la fecundación liberan sustancias que impiden que un segundo espermatozoide se fusione con el óvulo) y una serie de moléculas que van a ser necesarias durante el desarrollo temprano del embrión.
En la pubertad, bajo la influencia hormonal (estrógenos)à los ovocitos I reanudan la meiosisà cada 28 días un ovocito I (por lo general uno a la vez) completa la meiosis Ià obteniéndose dos célulasà primer cuerpo polar (que eventualmente se degenera) y el ovocito II (haploides=n). El ovocito II es mas grande ya que conserva casi todo el citoplasma.
El ovocito II inicia la meiosis II hasta detenerse en la metafase II, en espera de la fecundación.
En la ovulaciónà el ovocito II (detenido en metafase II) es liberado del ovario a las trompas de Falopio.
Si ocurre la fecundaciónà se completa la meiosis II obteniéndose dos célulasà el segundo cuerpo polar que se degenera y el ovulo (células haploides=n).
Espermatogénesis
Ocurre en los testículos.
Células primordialesà espermatogonias (2n)à células germinales.
En la pubertad por influencia hormonal (andrógenos) las espermatogonias se dividen por mitosis hasta alcanzar un número adecuado.
Algunas espermatogonias duplican su ADN y se diferencian en espermatocitos I (2n).
Los espermatocitos I comienzan la meiosis I y cuando termina se obtienen 2 célulasà espermatocitos II (haploides=n).
Los espermatocitos II comienzan la meiosis II produciendo cuatro célulasà espermátidas (n).
Las cuatro espermátidas por un proceso de maduración y diferenciaciónà espermatogénesisà se transforman en espermatozoides (n).
Las espermatogonias están conectadas por puentes citoplasmáticos ya que durante su división no se completa la citocinesis (división del citoplasma)à durante toda la división meiótica las células permanecen conectadas a través de estos puentes citoplasmáticos.
En el final de la diferenciación los espermatozoides individuales son liberados.
El proceso de diferenciación de una espermatogonia en cuatro espermatozoides tarda aprox. 72 días.
VIRUS
Sistemas macromoleculares que tienen capacidad de duplicarse.
En el nivel de organización se encuentran en un limbo entre lo vivo y lo no vivo.
No son células.
No tienen metabolismo propio.
Son agentes filtrables.
Son parásitos intracelulares obligatoriosà dependen de la maquinaria de una célula huésped para su replicación.
No pueden incorporar materia, transformar energía ni fabricar proteínas independientemente de la célula huésped.
Tienen un solo tipo de acido nucleícoà ADN o ARN, no ambos.
Estructura de los virus:
Virus desnudosà el acido nucleíco (genoma) está rodeado por proteínas llamadas capsómeros que en conjunto forman una cápside o nucleocápside.
Virus envueltosà la cápside está cubierto por una envolturaà hecha de fosfolipidos que el virus le roba a la célula que infecta, y proteínas propias del virus.
Dentro de la cápside está verdaderamente el virusà el coreà es el acido nucleico viral (lineal, circular, fragmentado, de doble cadena o de cadena simple) mas proteínas virales.
Ciclo de replicación viral
Ciclo lítico
Reconocimiento y adsorciónàel virus reconoce a la célula y se adhiere a ellaà la proteína viral interacciona con el receptor de la membrana celular.
Penetraciónà el virus inyecta su acido nucleicoà solo ingresa el genoma viral (sin cápside).
Eclipseà cada partícula del virus que ingrese a la célula se va a desarmarà dejan de verse. El virus pone a trabajar toda la maquinaria de la célula para élà se hacen copias de cada parte del virusà momento en que la célula gasta más energía.
Maduraciónà cuando las copias son suficientesà las partes del virus se ensamblanà se forman las nuevas partículas del virusà y salen de la celula:
En virus desnudosà las partículas hacen un brote (se llevan un pedacito de membrana). Si el brote es lentoà la célula llega a recuperar su pedacito y se salva. Si el brote es rápidoà la célula no llega a recuperarse y muere.
En virus envueltosà lisis celular (ruptura de la célula)à rompen la membrana y salen.
Ciclo lisogénico
Hay reconocimiento y adsorción, y hay penetración.
Antes de la etapa eclipseà el virus (ADN) migra hacia el núcleo y se inserta en el genoma celular. La célula tiene en su interior ADN del virusà si se divide va a replicar también el virus.
Pro-virusà cuando el genoma viral esta en el genoma de la célula.
En algún momento el virus va a salir del estado de lactancia y el ciclo lisogénico se convierte en lítico.
HIVà pertenece a los retro-virusà tienen ARN como único acido nucleíco y una enzima retro-transcriptasaà realiza la retro-transcripción.
En el core del virus esta el ARN donde la retro-transcriptasa puede convertir el ARN en ADN.
La proteína que reconoce el HIVà CD4à es de los linfocitos que amplifican la respuesta inmunitaria. Los linfocitosà se transforman en macrófagosà destruyen los virus pero se quedan con una parte y la exponen en su membrana.
EVOLUCION
Lamarckà propone una teoría de la evolución. Hace una serie de observacionesà observa que el medio ambiente cambia a lo largo del tiempo y que las especies evolucionan con él.
Define la evolución como un proceso lento y gradual.
Plantea à medio ambiente cambia y las poblaciones padecen estos cambios y como respuesta cambian sus comportamientos. *Ejemplo de la jirafa*. El uso intenso de unas partes del cuerpo produce su desarrollo, y el desuso que no se desarrollen. El carácter adquirido se transmite a los descendientes.
Ambiente cambiaà poblaciones responden modificándoseà interpretación intuitiva.
Evoluciónà camino a la perfecciónà proceso ortogenéticoà tiene un fin determinado.
Darwinà llega a las mismas conclusiones que Wallace.
1859à el origen de las especiesà reconoce que hay una relación entre la evolución y los cambios ambientales.
Nacen muchas mas crías de las que llegan a adultos.
Hay una competencia por los recursos limitados.
No todos los adultos se reproducen de la misma manera.
Si los recursos no fueran limitados la progresión seria geométrica.
Consiguen más recursos los que están más adaptados al ambiente.
Los que tienen características más adaptativas tienen más probabilidad de sobrevivir y reproducirse.
No pudo explicar el origen de la variabilidad.
Teoría sintética de la evolución
Genética de poblacionesà descarta la herencia de los caracteres adquiridos y explica el origen de la variabilidad a través de recombinación y mutaciones. Recombinar aquello que ya existeàrecombinar nuevos alelos.
Genética sistemáticaà ya no se considera como unidad evolutiva a la poblaciónà lo que cambia de la evolución son las frecuencias alelicas.
La evolución se puede interpretar como cambios en la genética de las poblaciones.
Ecuación de Hardy-Weinbergà proporciona un punto de referencia para medir los cambios de las frecuencias de alelos que siempre se producen en las poblaciones naturales.
Comportamiento de una población ideal:
NO mutaciones.
NO migraciones.
Población grande
Apareamientos al azar.
NO ventajas reproductivasà todos los alelos son igualmente viables.
Características de una población que no evoluciona.
Un conjunto de individuos de la misma especie, que comparten genes y comparten el mismo lugar y tiempoà son individuos que pueden reproducirse entre sí.
Especiación alopátricaà especiación fuera del lugar de origen. Consiste en la separación geográfica de una población de la misma especie, de forma que lleguen a producirse 2 o más poblaciones geográficamente aisladas que debido las condiciones ambientales del sector geográfico ocupado han ido evolucionando hasta formar nuevas especies.
Especiación simpátricaà formación de una nueva especie sin que haya barreras geográficas.
Mecanismosà poliploidíaà se da origen a un individuo tetraploide (4n)à es capaz de reproducirse sexualmente, pero quedará aislado genéticamente de la especie progenitora.
àhibridacionà se da origen a un individuo descendiente de padres pertenecientes a distintas especiesà suelen ser estériles.
Mecanismos de aislamiento reproductivo:
Ecológicoà individuos que ocupan el mismo territorio pero viven en diferentes hábitatsà no tienen la oportunidad de cruzarse.
Estacionalà especies que se reproducen en diferentes estaciones del año.
Etológico o conceptualà la atracción entre machos y hembras es necesaria para la unión sexual.
Mecánicoà diferencias en le copula en individuos de diferentes especies, dif. de altura o tamaño de los genitales.
Gaméticoà las gametas de un individuo de una especie no son reconocidas por el organismo de otro individuo de diferente especie.
Cigóticoà inviabilidad, esterilidad o reducción de ambos.
Hibrido estérilà el hijo es estéril.
Paleontologíaà sostiene que el registro fósil es compatible con un proceso evolutivo lento y gradual, guiado por la selección natural.
La teoría sintéticaà Neo-Darwinista acepta todos los postulados de Darwin excepto:
Explica el proceso evolutivo.
Sostiene que la clave del proceso evolutivo es la clave del aislamiento reproductivo.
Lento y gradual.
Los grandes cambios evolutivos son consecuencia de la sumatoria de pequeños cambios adaptativos.
Teoría saltacionista o de los equilibrios puntuados
Edredge y Gould dos paleontólogos que descubrieron que el registro fósil no muestra ninguna gradualidad y plantearon que la evolución es un proceso que se da a los saltos.
Mostrar que los procesos evolutivos de gran amplitud no pueden explicarse por la simple acumulación de procesos a pequeña escala, sino que responden a otras leyes y patrones.
El hombre y el resto de los primates alguna vez tuvimos un ancestro en común.
Evolución del desarrolloà estudia todos los mecanismos que alteran el desarrollo.
Teoría neutralistaà dice que la evolución es un fenómeno azarosoà no responde a ningún mecanismo de ningún tipo. Basado en un solo mecanismoà deriva genéticaà migración de un pequeño grupo de una población que se instala en otro lugar.